Pour l’Organisation mondiale de la santé (OMS), une meilleure capacité de séquençage est une priorité face à l’arrivée de plusieurs variants. Toutefois les analyses génomiques du virus ne proviennent que d’une poignée de pays. La France étant à cet égard très largement à la traîne. Pourtant, « Améliorer la couverture géographique du séquençage est essentiel pour que le monde ait des yeux et des oreilles braqués sur les mutations du virus ».
Pour surveiller les modifications du coronavirus susceptibles d’aggraver la pandémie ou de rendre les vaccins moins efficaces, les scientifiques doivent séquencer son génome. Mais peu de pays effectuent et partagent cette surveillance laborieuse, complexe et nécessaire. Les experts s’inquiètent donc autant des mutations à risque qui passent sous les radars que de celles qu’ils peuvent repérer.
La publication de la première séquence génomique du SRAS-CoV-2 en janvier 2020, au tout début de la pandémie, a permis de l’identifier comme un nouveau coronavirus et commencer à développer des tests de diagnostic et des vaccins. Depuis, des dizaines de milliers de séquences ont été téléchargées sur des bases de données publiques, permettant de suivre les mutations avec une précision et une vitesse jamais atteintes auparavant.
« C’est la première fois que nous voyons comment un agent pathogène évolue à cette échelle », relève Ewan Harrison, directeur de la stratégie et de la transformation au COG-UK et membre du Wellcome Sanger Institute, où une grande partie du séquençage est effectuée. « Nous apprenons que ces mutations s’accumulent bien plus vite que nous ne le pensions ».
La part du lion de ces informations provient d’un seul pays : la Grande-Bretagne. Actuellement, le programme britannique séquence 10.000 génomes par semaine (environ 6% des cas connus en Grande-Bretagne) et l’objectif est de doubler ce chiffre. « Le Royaume-Uni a écrasé tout le monde », déclare Emma Hodcroft, épidémiologiste à l’Université de Berne et co-développeuse du projet international de suivi du virus, Nextstrain. Le Danemark, relève-t-elle, séquence et partage aussi régulièrement les données, mais les informations provenant de la plupart des autres pays sont, au mieux, sporadiques.
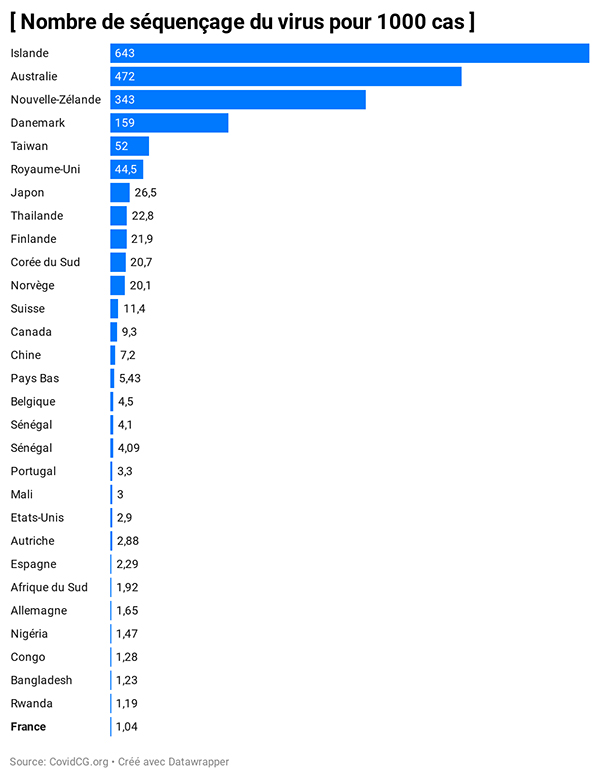
La France, quant à elle, est largement à la traîne. Elle fait partie des pays qui contribuent le moins à la recherche génomique internationale sur le virus. Alors que l’Islande effectue 643 analyses génomiques pour 1000 cas de Covid, l’Australie 472, la France se situe en bas de tableau avec un ridicule 1.04 pour 1000 cas. Moins que le Rwanda, la République du Congo ou le Bangladesh.
Identification des variants
Pourtant c’est grâce au séquençage que l’on a pu identifier des variants préoccupants en Grande-Bretagne, en Afrique du Sud et au Brésil.
Le nouveau variant à propagation rapide au Royaume-Uni est « comme une mini-pandémie au sein de la pandémie », dit le Dr Harrison. Sans surveillance systématique, les scientifiques n’auraient peut-être pas encore compris qu’il « change la donne ». L’alerte précoce n’a pas empêché sa propagation — des dizaines de pays l’ont détecté — mais elle a permis à d’autres de se préparer. Sans l’avertissement des scientifiques britanniques, le monde naviguerait à l’aveuglette, renchérit Emma Hodcroft.
D’autres variantes du virus ne sont devenues visibles que lorsqu’elles se sont répandues à l’international à partir de leur point d’origine. Ce mois-ci par exemple, une nouvelle souche, porteuse d’une mutation appelée E484K et dont les chercheurs craignent qu’elle puisse échapper à l’immunité, a été identifiée au Japon chez des personnes arrivant du Brésil.
Priorité au séquençage
Pour l’Organisation mondiale de la santé (OMS), une meilleure capacité de séquençage est une priorité.
Maria Van Kerkhove, responsable technique Covid-19 de l’OMS, a récemment qualifié d' »incroyable » le nombre de séquences partagées jusqu’à présent, mais déploré qu’elles ne proviennent que d’une poignée de pays. « Améliorer la couverture géographique du séquençage est essentiel pour que le monde ait des yeux et des oreilles (braqués) sur les changements du virus », a-t-elle déclaré.
Au départ, le nouveau coronavirus n’a pas montré beaucoup de diversité génétique, indique Emma Hodcroft, même s’il « a explosé à travers l’Europe ». « Nous avons pu voir qu’il semblait vraiment provenir de Chine, car toutes les séquences détectées dans le monde se retrouvaient dans les diverses séquences chinoises », précise-t-elle à l’AFP.
À l’été 2020, de nouvelles souches sont apparues, remplaçant les versions précédentes du virus. Les mutations font partie de l’évolution virale et se produisent lorsque le virus se réplique. Il s’agit « essentiellement de faute de frappe », explique la Dr. Hodcroft. La plupart des nouvelles souches ne confèrent aucun avantage au virus, certaines lui sont même défavorables. Mais parfois, une mutation augmente le caractère infectieux ou provoque une maladie plus grave.
Plus un virus infecte de gens, plus la possibilité de mutation est grande, et la probabilité augmente chez une personne dont le système immunitaire est chroniquement affaibli. C’est peut-être ainsi que le nouveau variant est apparu au Royaume-Uni, et les chercheurs séquencent maintenant les souches provenant de patients immunodéprimés, indique le Dr Harrison.
L’étape suivante consiste à étudier comment les différentes mutations affectent la transmission du virus, la gravité de la maladie et l’efficacité du vaccin, et à prédire le plus rapidement possible le comportement d’un nouveau variant. Pour l’OMS, le séquençage mondial aidera « à mieux comprendre le monde des agents pathogènes émergents et leurs interactions avec les humains et les animaux dans une variété de climats, d’écosystèmes, de cultures, de modes de vie etc. ».
Mais le séquençage à grande échelle est complexe sur le plan logistique. L’Institut Wellcome Sanger, au sud de Cambridge, en Angleterre, stocke les dizaines de milliers d’échantillons qu’il reçoit chaque jour dans d’énormes congélateurs et il a conçu toute une infrastructure robotique pour les trier et les utiliser dans ses recherches.
Mobilisation poussive en France
Ce n’est pas le cas en France. Le pays dispose pourtant selon Bruno Coignard, directeur des maladies infectieuses à Santé Publique France, de 46 laboratoires hospitaliers en capacité de faire du séquençage. Mais la mobilisation de ces équipements et des personnels compétents pour les faire tourner tarde. D’autant que tous ces équipements ne sont pas paramétrés pour réaliser des recherches en infectiologie sur des virus.
Pour répondre au besoin à court terme, « une trajectoire de renforcement des plateformes existantes leur permettant de remplir les nouveaux objectifs de surveillance et la mise en place progressive de nouvelles plateformes et des compétences associées doivent être définies ». Ces mots sages proviennent d’un avis du Conseil scientifique. Le problème est qu’ils datent du 12 janvier dernier seulement. Dans de nombreux autres pays, la stratégie de séquençage a été définie depuis le printemps 2020.
« Nous n’avons pour l’instant que le pouvoir d’écoper en urgence, car un vrai programme de surveillance n’a pas pu être mis en place à cause de l’urgence de la crise sanitaire » a ainsi confié au Figaro Agnès Delaunay-Moisan, microbiologiste au Centre de biologie intégrative et de génétique moléculaire du CEA.
La mobilisation progressive de toutes les ressources est essentielle pour accélérer la cadence de dépistage ; même si les objectifs affichés sont loin de rivaliser avec l’activité britannique où 10 % des prélèvements positifs sont séquencés, soit actuellement plus de 30 000 par semaine. Le gouvernement français projette, selon le Journal International de Médecine, de réaliser 6 000 séquençages par semaine en avril (ce qui sera peut-être un peu tard par rapport aux prévisions de diffusion du variant britannique) contre 1 500 aujourd’hui (moins de 1 % des infections repérées).
Face à une telle confusion, beaucoup déplorent l’absence de pilotage et de moyens. « Pour les financements, on nous a dit qu’il y aurait 10 millions d’euros qui seraient pour le séquençage. On l’espère, parce qu’il ne s’agit pas de faire un travail pour un mois ou deux » s’agace ainsi le généticien Philippe Froguel, sur Franceinfo. Ce dernier a fait appel au président de la République pour mettre en place un consortium de séquençage du virus Sars-CoV-2 à l’échelle nationale.
Le virologue et membre du Conseil scientifique Bruno Lina reconnaît, dans Libération, que les capacités de séquençage françaises ne sont pas à la hauteur de l’enjeu épidémique. Il admet que l’initiative du professeur Froguel a certainement « secoué le cocotier ».
Pour rattraper son retard et organiser du séquençage à grande échelle, la France qui prétend posséder des machines capables de séquencer et les compétences pour décrypter les résultats, mise sur une nouvelle agence, l’ANRS maladies infectieuses émergentes. « On peut aujourd’hui séquencer 1 500 génomes par semaine » précise le professeur Lina. « Si on bâtit sur l’existant, et qu’on complète nos ressources, à échéance d’avril, on peut être en capacité de séquencer 6 000 génomes par semaine, soit 300 000 génomes par an. C’est l’équivalent de ce qui a été fait à l’échelle mondiale en 2020 ».
Au doigt mouillé
En attendant que cette stratégie de séquençage se mette en place et que des directives opérationnelles soient données par les autorités, la France se contente du doigt mouillé ou pour être plus agréable, des modélisations, comme celle présentée la semaine dernière par l’inserm qui évoque une « possible » circulation majoritaire du variant anglais sur notre territoire en mars.
Savoir précisément comment circulent les différents variants est indispensable pour diriger une stratégie efficace de lutte contre le virus. « L’important, c’est de savoir combien il y en a sur le territoire français parce que ça va conditionner notre stratégie dans les trois mois qui viennent », a déclaré l’épidémiologiste et membre du Conseil scientifique Arnaud Fontanet à BFM TV.
Repérer tous les variants en circulation est une tâche d’une grande ampleur. Aujourd’hui, il y a quatre variants identifiés. Les spécialistes comme le professeur Bruno Lina ont une seule certitude : « le variant le plus transmissible va chasser la souche en place. Est-ce que ce sera le variant anglais, le sud-africain, le brésilien ou un autre ? Je suis incapable de le dire. On est dans une compétition entre virus. Celui qui aura la capacité de diffusion la plus forte l’emportera. Le variant anglais a un avantage de réplication et donc de transmission. Tout comme le variant sud-africain qui, en plus, semble avoir un petit avantage d’échappement immunitaire ». Pour lutter dans cette course aux variants, « Il faut augmenter nos capacités de séquençage, et collecter un échantillonnage de prélèvements suffisamment large pour pouvoir identifier un événement rare qui surviendrait. » pense le professeur Lina.
La mise en place d’un tel dispositif est cruciale dans la pandémie actuelle. Mais elle est fondamentale pour être en mesure, une fois cette épidémie terminée, de détecter l’arrivée de nouveaux pathogènes et éviter d’être pris au dépourvu comme nous l’avons déjà été.
Avec AFP